一 : 如下图,镐京与曲阜的关系是[]A.王都与封国都邑B.均为封国都邑C.王都与郡府D.均为郡府
| 如下图,镐京与曲阜的关系是 |
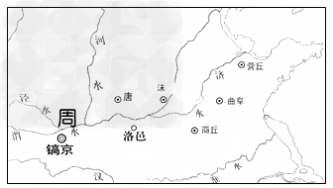 |
| [ ] |
| A.王都与封国都邑 B.均为封国都邑 C.王都与郡府 D.均为郡府 |
| A |
考点:
考点名称:周的分封制分封制:
分封制也称分封制度或封建制,即狭义的“封建”,由共主或中央王朝给王室成员、贵族和功臣分封领地,属于政治制度范畴。古代宗法制是分封制的基础,在家庭范围是为宗法制,在国家范围是为分封制。中国古代帝王分封诸侯的制度。周灭商和东征以后,曾分封同姓和功臣为诸侯,以为藩屏。诸侯的君位世袭,在其国内拥有统治权,但对天子有定期朝贡和提供军赋﹑力役等义务。
夏商周的政治制度发展: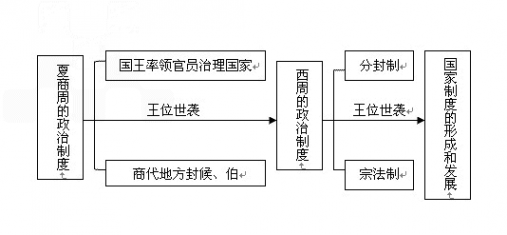
二 : 思维与大脑的魅力关系
曾经从一本书看到过,不只是哪本书,想法太多我开始有点犯糊涂,印象不是很深了。也许只有在这样的情况下,我才会意识到自己大脑的应用还不足,我才看了这本书一天,它就告诉了我,我的“思维导图|”并不清楚。没错,我记起来了,是《思维导图》。这种下意识的追踪,告诉了我的思维并没有那么糟糕。也许你一直在怀疑你的记忆力没有那么好,可能是因为你太笨,老是记不清。其实无需我给你找理由,只要你静下心来想想,只是你没有达到自己学习的量和那种学习的态度而已。如果你被我说中了,你没有努力过,或许我只说对一半,只是你半途而废,因为见效很慢。这些理由,这些因素,让我们自然而然的习惯的以为我们是“笨”的。错了,你错了,你之所以因为记不住东西而做不好事情,或者你根本不知道该怎么做,真相从来都不为没有准备的人而浮现。告诉你自己,你是不是很糊涂,比如你会糊涂的一片空白,无处着手。人很聪明,没有一个人是“笨”的,思维导图告诉我们,只要你有一个具备思考能力的大脑,你就能放射无数个事物,它是一个充满光芒的智慧球体。当我打开《思维导图》,我好奇而又兴奋的去翻阅着,我发现大脑其实就是一个思维导图,它被分成了思维逻辑上的左右脑,两脑控制着“节奏丶空间感丶格式塔丶想象丶白日梦丶色彩丶尺度丶词汇丶逻辑丶数字丶顺序丶线性感丶分析丶列单”这些不同的功能作用。人类大脑的五大功能分为接收丶保持丶分析丶输出和控制,保持其实就是一种记忆功能,分析是在有保持的功能上而运作的。那思维导图就是超控大脑的最有帮助的表现形式。尽管我们知道了这些,大脑的合理分配使用,实施起来却是宇宙的最大难题。爱因斯坦死前给自己的定义是我这一身的价值也只是利用了还不到百分之一的脑细胞,对,这不是谦虚,这是一个事实。人类的巨大潜能在告诉我们什么?给自己一句鼓励的话和一句自信的话,我一定要成为一个有智慧的人,我一定是有智慧的人。(没有人是笨蛋,只是自己意识不到,也懒得去开发自己的潜力)
三 : 学生自身素质的发展与美术教学的关系
我们大家都知道,每个学生都是一个特殊的个体,在他们身上既体现着发展的共同性特征,又表现出巨大的个体差异,因此,就有了所谓的“好生”与“差生”之说。然而,因为美术学科的独特性,我们决不能简单地评价一个学生的“好”与“差”。记得有位专家说过:在美术课中没有差生!的确,每个人的艺术天赋有所差距,但表现艺术的机会都是均衡的。我们应立足于学生的个体差异,发现、发展学生多方面的潜能,在遵循共同规律对学生进行教育教学的同时,实施区别指导和分层教学,正确评价每一个学生的发展。他的作用致关重要。因此,现代教育要求教师必须在新的教学理念的指导下,正确看到评价这一功能,充分发挥评价的作用,逐步形成以学生为主的主体性评价。四 : 小麦生长素结合基因TaABP1-D的克隆、功能标记开发及其与株高的关联
作物学报 ACTA AGRONOMICA SINICA 2012, 38(11): 2034?2041 http://www.chinacrops.org/zwxb/ ISSN 0496-3490; CODEN TSHPA9 E-mail: xbzw@chinajournal.net.cn DOI: 10.3724/SP.J.1006.2012.02034
小麦生长素结合基因TaABP1-D的克隆、功能标记开发及其与株高的关联
乔麟轶1,** 张 磊1,** 张文萍1 赵光耀2 王 玺1,* 贾继增2,*
12沈阳农业大学农学院, 辽宁沈阳 110866; 中国农业科学院作物科学研究所 / 农作物基因资源与基因改良国家重大科学工程 / 农业部作物基因资源与种质创制重点实验室, 北京 100081
摘 要: 生长素在植物生长发育过程中通过建立浓度梯度来调控植物的株型。[www.61k.com)ABP1 (auxin binding protein)作为生长素受体, 在质膜上相关生长素响应活动中起重要作用。本研究从普通小麦中国春基因组数据库中分离了TaABP1基因的基因组序列, 根据基因组间序列差异将 TaABP1 定位于小麦第5同源群。在中国春中克隆得到TaABP1-D基因的gDNA和cDNA序列。TaABP1-D基因的开放阅读框为1 887 bp, 编码205个氨基酸, 含有ABP1蛋白典型的内质网滞留信号KDEL及Box区域。表达分析表明, TaABP1-D在普通小麦中国春拔节期的根、茎基部、茎上部和叶尖均有表达, 相对表达量为叶尖>茎上部>根>茎基部, 与小麦的株型发育关系密切。系统发育分析表明, ABP1基因在植物中较为保守, TaABP1-D是水稻OsABP1的直向同源基因。针对TaABP1-D基因上游调控区重复序列差异(GT)6/5开发了一个SSR标记, 该标记在W7984×Opata85重组自交系(RIL)群体中对株高的表型变异解释率为9.7%, 是一个与株高极显著关联的功能标记; 其中对应高秆类型的等位变异属野生种特有, 在栽培种中被淘汰, 推测TaABP1-D基因在小麦驯化过程中可能经历了瓶颈效应。
关键词: 小麦; TaABP1-D基因; 同源克隆; 表达分析; 功能标记; 相关性分析
Molecular Cloning and Development of a Functional Marker of TaABP1-D Gene Associated with Plant Height in Bread Wheat
QIAO Lin-Yi1,**, ZHANG Lei1,**, ZHANG Wen-Ping1, ZHAO Guang-Yao2, WANG Xi1,*, and JIA Ji-Zeng2,* 1 College of Agronomy, Shenyang Agricultural University, Shenyang 110866, China; 2 National Key Facility for Crop Gene Resources and Genetic Improvement / Key Laboratory of Crop Gene Resources & Germplasm Enhancement, Ministry of Agriculture / Institute of Crop Sciences, Chinese Academy of Agricultural Sciences, Beijing 100081, China
Abstract: AUXIN BINDING PROTEIN1 (ABP1) is one of the ?rst characterized proteins that binds auxin. It ful?lls the criteria for an auxin receptor and plays an important role in auxin responses on the plasma membrane. In this study, the genomic sequence of a novel gene, designated TaABP1, was isolated from the hexaploid wheat (Triticum aestivum L.) Chinese Spring genome data-bases. The result of chromosomal location showed that TaABP1 was on homoelogous group 5 of chromosomes in wheat. Then TaABP1-D was cloned in Chinese Spring. Sequence analysis showed that the complete open reading frame (ORF) of TaABP1-D was 1887 bp in length, encoding a putative protein composed of 205 amino acids with the endoplasmic reticulum retention se-quence Lys-Asp-Glu-Leu (KDEL) and box regions. The expression of TaABP1-D was detected in root, basal stem, upper stem, and leaf tip of wheat at jointing stage with the expression levels in the order of leaf tip > upper stems > root > base stem. Sequence alignment proved that TaABP1-D shared high homology with rice OsABP1 (82% protein sequence identity) and other plant ABPs reported. Using a population of recombinant inbred lines from the cross between W7984 and Opata85, an SSR marker was devel-oped based on (GT)6/5 divergence in the upstream sequence of TaABP1-D. This marker proved to be highly correlated with plant height, and the phenotypic variation explained was 9.7%. The W7984 type allelic variation for tall plant was specific in wild va-
本研究由国家转基因生物新品种培育科技重大专项(2011ZX08009-001), 教育部博士点专项基金(20092103120010)和辽宁省科技厅博士启动基金(20091074)资助。
* 通讯作者(Corresponding authors): 王玺, E-mail: wxi2000@126.com; 贾继增, E-mail: jzjia@mail.caas.net.cn
同等贡献(Contributed equally to this work) **
第一作者联系方式: E-mail: qiaoly1988@126.com (乔麟轶); E-mail: lnzhanglei2003@yahoo.com.cn (张磊)
Received(收稿日期): 2012-03-22; Accepted(接受日期): 2012-06-15; Published online(网络出版日期): 2012-09-10. URL:
abp138 小麦生长素结合基因TaABP1-D的克隆、功能标记开发及其与株高的关联
第11期
乔麟轶等: 小麦生长素结合基因TaABP1-D的克隆、功能标记开发及其与株高的关联 2035
rieties, and eliminated in cultivated varieties. This indicates that the favorable variation of TaABP1-D has been fixed in cultivated varieties of wheat, and a bottleneck effect might occur during the domestication of wheat.
Keywords: Wheat; TaABP1-D; Homologous cloning; Expression analysis; Functional marker; Association analysis
生长素是最早发现的一类植物激素, 它在调控植物的生长发育以及对环境的响应过程中起重要作用[1]。[www.61k.com)生长素受体的研究对于揭示生长素信号转导的途径、深入了解生长素对植物诸多生理过程的作用机制有重要意义。目前, 生长素受体除已知的TIR1/AFBs[2-3]和新近发现的相位激酶关联蛋白SKP2A[4]外, 生长素结合蛋白ABP1 (auxin binding protein)在质膜上的快速反应中也作为一种生长素受体存在[5]。
61阅读提醒您本文地址:
1972年首次在玉米胚芽鞘膜上发现ABP1[6]。它是一种在植物中广泛存在的低丰度蛋白, 结构较为保守, 开花植物典型的ABP1包含N端一个用来进入分泌途径的信号肽序列和C端一段具有KDEL特征的内质网滞留序列[7], 大多位于内质网腔, 也有少量存在于质膜上或附近和细胞壁中, 但是只有位于质膜的ABP1才有生长素结合能力。对拟南芥和玉米等模式作物的研究表明, ABP1在调节细胞周期[8]、细胞扩增[9-10]和细胞骨架重组[11]等质膜快速反应过程中发挥重要作用。
近来研究还发现ABP1能诱导细胞对PIN蛋白的内吞作用[12], 用一个ABP1δKDEL与突变体进行ABP1的过分泌表达, 发现细胞外的ABP1促进了细胞网格蛋白对PIN蛋白的内吞作用, 施加生长素后促进作用被抵消。而另一株不能结合生长素的abp1-5突变体苗[11]在施加生长素后则不能抵消这种促进作用。在另一个实验中也发现ABP1与生长素结合能阻止细胞对PIN蛋白的内吞, 导致质膜上大量PIN的富集[13]。这表明ABP1与生长素相结合抑制了ABP1对PIN蛋白的内吞作用。
目前虽然已克隆了一些植物ABP1基因[14-15], 但尚未见有小麦ABP1基因的报道。小麦基因组庞大且复杂, 其研究远远落后于拟南芥、水稻等模式植物, 利用其他物种已有的基因序列信息进行同源克隆是分离克隆小麦功能基因的有效手段。本实验采用该方法得到小麦ABP1-D基因完整编码序列, 通过生物信息学分析获知该基因的一般特征、蛋白结构, 并利用RT-qPCR研究TaABP1-D在小麦拔节期不同组织器官的表达特异性; 针对多态性位点开发功能标记, 进行标记与性状的关联分析和等位变异分析, 以期为利用该基因改良小麦农艺性状奠定
基础。
1 材料与方法
1.1 材料
普通小麦(Triticum aetivum L.)品种中国春用于基因克隆; 中国春缺体-四体材料用于染色体定位; 从小麦微核心种质中选出101份栽培种, 外加37份野生种(人工合成小麦)组成具有广泛遗传代表性的138份小麦种质资源, 用于基因多样性分析; W7984 ×Opata85 RIL国际作图群体由国际小麦作图组织(ITMI)培育, 共有114个株系。上述材料均由中国农业科学院作物科学研究所提供。
采用Devos等[16]改进的酚–氯仿法提取小麦基因组DNA。按RNA提取试剂盒(天为时代公司, 北京)说明书的方法提取总RNA, 用反转录试剂盒(M-MLVRT, Invitrogen, 上海)反转录得到小麦cDNA。
1.2 TaABP1基因序列的分离和染色体定位
根据已知的水稻OsABP1基因cDNA开放阅读框(GenBank登录号为DQ538525)对小麦EST库(http://compbio.dfci.harvard.edu/tgi/)进行Blast检索, 得到同源EST序列TC405894, 长度为818 bp。将该EST序列提交小麦基因组数据库(http://www.cere-alsdb.uk.net/)进行搜索, 获得103条基因组序列片段, 经拼接整理得到小麦ABP1基因的基因组contig 528059/382946。
根据基因组间序列差异设计引物SF1/SR1 (表1), 利用中国春缺体-四体材料对TaABP1进行染色体定位。
1.3 TaABP1-D基因的克隆和测序
将contig528059/382946-D与OsABP1基因cDNA序列比对, 界定小麦基因编码区, 跨越开放读码框设计引物gF/gR (表1), 分别以普通小麦中国春基因组DNA和cDNA进行PCR扩增。扩增产物经电泳后回收纯化, 选用平末端连接试剂盒(全式金公司, 北京)连接转化, 载体为pEASY-T1, 感受态细胞为TOP10。采用碱裂解法提取经筛选得到的阳性克隆质粒DNA, 按照BigDyeR Terminator v3.1 Cycle Sequencing Kit方法、使用3730 XL DNA Analyzer (Applied Biosystem Inc., 美国)测序。
abp138 小麦生长素结合基因TaABP1-D的克隆、功能标记开发及其与株高的关联
2036
作 物 学 报
表1 本研究设计的TaABP1基因的引物
Table 1 Primers of TaABP1 gene designed in this study
第38卷
引物序列 退火温度 引物组合 延伸时间 产物长度
Sequence (5'→3') Primer pair Elongation time (s) Product size (bp) Annealing temperature (°C)
SR1: ATAAACAAAATGCACCAGAG
gF/gR gF: CACGCTGGAGCGTCACTAT 55 (gDNA), 62 (cDNA) 180 (gDNA), 60 (cDNA) 2340 (gDNA), 650 (cDNA)
gR: GCTGCTTCATGCTCTCAAGT
qF/qR qF: GCCGTGAACTTTATTTGGTG 59 70 1866
qR: ACAATGAATAGTGACGCTCC
qSF1/qSR1 qSF1: CAGGCGCTGTTCTAGGGTT/ 58 30 182180
qSR1: CACCGCCTGTAAAACTGGAT
RtF/RtR RtF: CCTTCCTGCCCCCGAGACAA/ 58 30 102
RtR: CGCGCCCGCAACGGTTATAT
1.4 生物信息学分析
采用DNAStar中的SeqMan进行序列组装、拼接和比对分析, 并用Editseq查找全长序列的开放阅读框(ORF), 将其翻译成氨基酸序列。(www.61k.com)水稻及其他物种的ABP1基因序列来自NCBI数据库(http://www. ncbi.nlm.nih.gov/), 将核酸序列提交到GenBank进行Blastn分析。蛋白比对和系统发育分析在ClustalX(1.83)软件中完成, 用MEGA4软件构建系统发育树。
采用ProtParam工具(http://www.expasy.org/tools)计算分子量、等电点和蛋白亲/疏水性。利用Smart服务器(http://smart.embl-heidelberg.de/)在线分析保守域和功能位点。用NPSA工具(http://npsa-pbil.ibcp. fr/cgi-bin/secpred_sopma.pl)分析TaABP1-D二级结构。用SWISS MODEL程序(http://au.expasy.org/tools)分析TaABP1-D的三级结构。
1.5 TaABP1-D基因的组织表达分析
取中国春拔节期的根、茎基部、茎上部和叶尖组织提取总RNA, 根据TaABP1-D cDNA序列设计引物RtF/RtR (表1), 对TaABP1-D基因进行RT-qPCR分析, 以小麦Tubulin为内标基因。
1.6 TaABP1-D的功能标记开发及关联分析
以亲本组合W7984/Opata85为模板, 用引物qF/qR (表1)对TaABP1-D编码区上游序列进行扩增, 产物回收测序。根据亲本间序列差异开发分子标记, 并对W7984×Opata85 RIL F6群体的114个家系进行
PCR扩增, 用6%变性聚丙烯酰氨凝胶电泳及银染检测扩增产物(65 W恒功率)。记录电泳带型, 使用SPSS 11.0统计软件对不同等位变异所对应的表型平均值进行t检验[17]。
1.7 TaABP1-D等位变异频率分析
利用开发的标记检测138份中国小麦种质资源的TaABP1-D位点, 按野生种、农家种和育成种3类分别统计等位变异的比率和分布。
61阅读提醒您本文地址:
2 结果与分析
2.1 小麦TaABP1基因的染色体定位
从小麦基因组数据库中分离得到的TaABP1基因组序列contig528059/382946含有3种差异(图1), 推测这3种差异序列可能是TaABP1基因来自3个基因组的不同拷贝, 据此设计引物SF1/SR1在中国春缺体-四体中定位该基因, 由于扩增产物的长度差异, 可以分离出487、475和465 bp基因组特异带型, 它们被定位在小麦第3同源群上(图2)。
2.2 小麦TaABP1-D基因的克隆
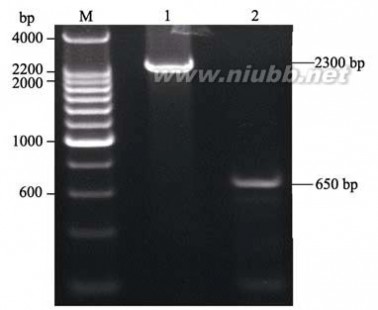
使用基因全长引物gF/gR扩增普通小麦中国春基因组DNA和cDNA, 分别得到2 300 bp和650 bp的目标片段(图3), 经测序证实均含有目标EST序列TC405894; 通过比较2个目标片段序列, 明确TaABP1-D基因含有5个外显子和4个内含子, ORF全长618 bp, 编码205个氨基酸(图4), 理论分子量为22.3 kD, 等电点为6.29。

图1 小麦TaABP1基因组序列存在3种长度差异
Fig. 1 Genomic sequence of TaABP1 contained three fragments different in length
abp138 小麦生长素结合基因TaABP1-D的克隆、功能标记开发及其与株高的关联
第11期

乔麟轶等: 小麦生长素结合基因TaABP1-D的克隆、功能标记开发及其与株高的关联 2037
位含有ABP1基因家族的保守域(图5)。(www.61k.com)蛋白序列分析表明, TaABP1-D与OsABP1的序列一致性达82%; TaABP1-D蛋白序列包含ABP1蛋白家族的所有特征, 即N端信号肽序列、BoxA、BoxB、BoxC区和ABP1蛋白末端KDEL特征区(图6), 证明所克隆的
图2 TaABP1的染色体定位
Fig. 2 Chromosomal localization of TaABP1
1: Chinese Spring; 2: H2O; 3: N5AT5D; 4: N5AT5B; 5: N5BT5D;
6: N5BT5A.
基因为ABP1基因。蛋白疏水性分析表明, N端具有17个氨基酸残基的疏水性信号序列, 利于ABP1在内质网膜间的穿透和转移, 起信号转导作用; BoxA区被认为是主要的生长素结合位点; C末端特征区的KDEL为内质网滞留信号, 暗示此多肽链可能位于内质网上。
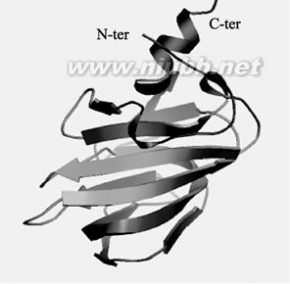
二级结构预测结果显示TaABP1-D蛋白含有丰富的β折叠; 从预测的三维结构可以看出TaABP1-D蛋白在C端有一段α螺旋结构, 此外由β片层结构形成一个疏水口袋, 组成cupin保守结构域[18](图7)。该结构域对于生长素的结合有重要意义。
图3 引物gF/gR在中国春中的扩增图谱
Fig. 3 Amplification profile of primer pairs gF/gR in Chinese
Spring
M: 200 bp DNA ladder; 1: gDNA; 2: cDNA.
2.3 普通小麦TaABP1-D 基因序列分析
对TaABP1-D编码的氨基酸序列进行结构和功能位点分析, 结果显示在TaABP1-D蛋白1~36位含有一个36个氨基酸长度的信号肽序列, 第41~206
2.4 ABP1 蛋白的系统发育树
选取13种典型植物的ABP1蛋白序列, 用ClustalW软件将之与TaABP1-D进行序列多重比对, 发现TaABP1-D与其他物种ABP1序列相似性普遍较高, 与亲缘关系较近的大麦和燕麦的序列相似性分别为92%和85%, 与苎麻、葡萄和陆地棉等物种的序列相似性也达到59%以上。在进化上小麦ABP1与单子叶植物处在一个进化分支, 与苎麻、葡萄等双子叶植物的进化关系较远(图8)。由此推断, ABP1基因的分化与物种的分化可能是同时发生的。
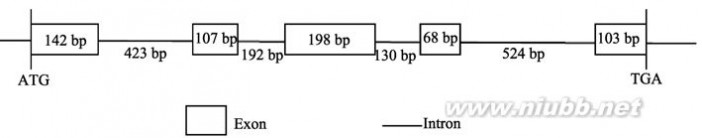
图4 TaABP1-D基因结构
Fig. 4 Exon-intron structure of TaABP1-D gene

图5 TaABP1-D编码的氨基酸序列的结构及功能位点
Fig. 5 Structure and function site in the amino acid sequence
deduced from TaABP1-D
2.5 TaABP1-D基因的表达模式
TaABP1-D在小麦根、茎基部、茎上部和叶尖均有表达, 相对表达量为叶尖>茎上部>根>茎基部(图9), 可见TaABP1-D在生长旺盛的部位表达量较高, 反映该基因与植物生长发育关系密切。
2.6 TaABP1-D功能标记的开发及其与株高的关联分析
分析W7984和Opata85中的TaABP1-D序列,
abp138 小麦生长素结合基因TaABP1-D的克隆、功能标记开发及其与株高的关联
2038
作 物 学 报 第38卷
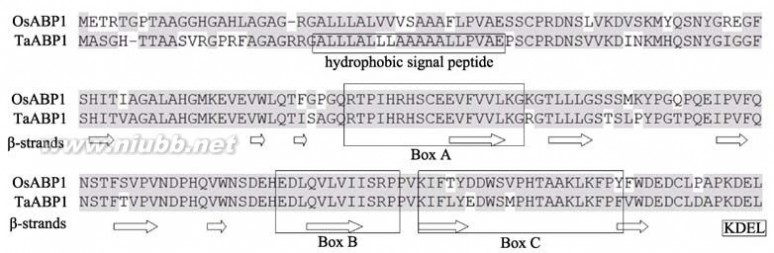
图6 OsABP1和TaABP1-D的蛋白序列比对
Fig. 6 Protein sequence alignment of OsABP1 and TaABP1-D
(图11)。(www.61k.com]
利用群体基因型和表型数据, 对RIL群体株高、穗长、穗粒数、千粒重等重要农艺性状进行单标记分析, 结果表明Opata85带型和W7984带型的植株株高存在极显著差异(t = 3.106, P < 0.01), Opata85带型对应较矮株高(109.05±1.71 cm), W7984带型对应较高株高(115.98±1.40 cm), qSF1/qSR1与株高显著关联(P = 0.0025), 表型变异解释率达9.7%。因此,
TaABP1-D基因很可能参与调控小麦株型发育。
图7 SWISS模型预测的TaABP1-D蛋白三级结构
Fig. 7 Tertiary structure of TaABP1-D predicted by SWISS
MODEL
2.7 TaABP1-D基因的等位基因分布与多样性分析
利用标记qSF1/qSR1检测138份小麦种质资源, 发现W7984带型只存在于野生种Am6、Am9、96S-224和96S-225中, 分布频率为10.8%; Opata85带型在农家种和育成种中的分布频率达100%, 没有W7984带型出现(表2)。
61阅读提醒您本文地址:

发现二者在ATG上游?336 bp处存在一个(GT)6/5的重复序列差异(图10), 由此开发了SSR标记qSF1/ qSR1。该标记在W7984×Opata85 RIL群体中可扩增出2种带型, 其长度分别是182 bp和180 bp
图8 ABP1蛋白的系统发育分析
Fig. 8 Phylogenetic analysis of ABP1 proteins
abp138 小麦生长素结合基因TaABP1-D的克隆、功能标记开发及其与株高的关联
第11期

乔麟轶等: 小麦生长素结合基因TaABP1-D的克隆、功能标记开发及其与株高的关联 2039
核苷酸序列多态性分析表明, TaABP1-D基因在农家种和育成种中非常保守, 核苷酸多态性均为0, 而在野生种中多态性值(π)为0.21, θ值、Hd值、k值均呈现出相同的趋势, 说明TaABP1-D基因在从野生种到农家种的驯化过程中可能经历了人工选择, 是一个驯化相关基因(表3)。[www.61k.com)
3 讨论
图9 小麦拔节期不同组织TaABP1-D基因的表达水平 Fig. 9 Relative expression levels of TaABP1-D gene in
different tissues of wheat
生长素在组织发育过程中通过建立浓度梯度来调控植物的株型[19], 而PIN蛋白作为生长素输出载体[20], 在建立和维持生长素梯度中起重要作用, 最终影响植物形态[21]。由于ABP1与生长素结合可以

图10 TaABP1-D编码区上游?336 bp处在W7984和Opata85间存在(GT)6/5重复序列差异
Fig. 10 Sequence difference of (GT)6/5 at the ?336 bp site of TaABP1-D

between W7984 and Opata85
图11 SSR标记qSF1/qSR1在W7984×Opata85 RIL群体中的分离 Fig. 11 Segregation of SSR marker qSF1/qSR1 in W7984 × Opata85 RILs
箭头分别指示父本带型(182 bp)和母本带型(180 bp)。O: Opata85; W: W7984; 1~25: RIL群体单株。
The arrows show the banding patterns of male parent (182 bp) and female parent (180 bp). O: Opata85; W: W7984; 1–25: individuals from
the RIL population.
表2 qSF1/qSR1标记位点2种等位变异在138份小麦种质资源中的分布
Table 2 Distribution of two alleles on locus qSF1/qSR1 in 138 entries of wheat accessions 种质资源类型 Type of germplasm 野生种 Wild species 农家种 Landrace 育成种 Modern cultivar
表3 TaABP1-D在野生种、农家种和育成种中的序列多态性分析
Table 3 Analysis of sequence diversity of TaABP1-D in modern cultivars, landraces, and wild species
种质资源类型 Type of germplasm 野生种 Wild species 农家种 Landrace 育成种 Modern cultivar
单倍型
Haplotype h1, h2
π (×103) 0.21
θ (×103) 0.26
Hd 0.198
k 0.396
总份数 Total accessions
Opata85带型 Opata85 banding pattern 份数 Accession
百分率 Percentage (%)
W7985带型 W7985 banding pattern 份数 Accession
百分率 Percentage (%)
10.8 0 0
37 33 89.2 4 0 0
50 50 100.0 51 51 100.0
h2 0.00 0.00 0.000 0.000 h2 0.00 0.00 0.000 0.000
h1: W7984类型; h2: Opata85类型。π: 核苷酸多态性; Hd: 单倍型多态性; k: 核苷酸差异平均值。
h1: W7984 type; h2: Opata85 type. π: nucleotide diversity; Hd: haplotype diversity; k: average number of nucleotide diversity.
abp138 小麦生长素结合基因TaABP1-D的克隆、功能标记开发及其与株高的关联
2040
作 物 学 报 第38卷
调节细胞对PIN蛋白的内吞作用进而影响生长素的极性运输, 因此可将ABP1基因作为株型改良的候选基因。[www.61k.com)本研究通过RT-qPCR证明, TaABP1-D在小麦拔节期的叶尖和茎上部等生长素活动旺盛的部位活跃表达, 说明TaABP1-D与小麦株型发育关系密切。
我们曾对10份小麦材料中的TaABP1-D基因进行序列比较, 发现编码区序列完全相同, 说明该基因高度保守。对TaABP1-D编码区上游测序, 根据亲本间序列差异在?336 bp位置开发了一个SSR标记, 该标记与W7984×Opata85 RIL群体的株高呈极显著相关, 可解释9.7%的表型变异。在TaABP1-D基因上游序列?538 bp和?408 bp位置发现各有一个顺式作用元件GCbox (GGGCGG), 其下游序列30~110 bp处为转录起始点(图10)。因此, 该SSR标记位点应该位于TaABP1-D基因的5'端非翻译区(5'-un- translated region, UTR), 该区域中重复序列的变异可能导致转录起始位点的改变从而对基因的表达产生影响[22]。初步推断TaABP1-D基因可能在转录水平调控株型发育, 这有待进一步验证。我们将对不同等位变异转录本深入分析, 可能存在不同的转录起始位点、基因表达差异和转录效率的差异。
多样性变化(通常是多样性降低)是检测作物驯化与品种改良过程中人工选择位点的主要方法之 一[23]。小麦微核心种质(mini-core collection, MCC)代表了70%多样性, 是分析多样性的理想群体[24-25]。本研究从小麦微核心种质中选取了具有代表性的101份栽培种, 再加上37份野生种和人工合成小麦组成具有广泛遗传代表性的分析样本, 通过对TaABP1-D基因在野生种与栽培种中的多样性分析, 发现其在栽培种中的多样性显著下降(栽培种为0), 推测该位点可能在驯化过程中经历了人工选择。连锁群体中发现TaABP1-D的野生型株高显著高于栽培型, 而株高是一个重要的驯化相关性状
61阅读提醒您本文地址:
[26-27]
, 这
一结果进一步表明该基因可能是一个驯化相关基因。最新的研究还发现ABP1在拟南芥中与花期密切相关
[28]
, 假如TaABP1-D一因多效, 可以控制小
麦花期, 将有可能通过该基因改良小麦性状。
4 结论
根据水稻OsABP1基因同源比对获得普通小麦TaABP1基因组序列, 根据基因组间序列差异将TaABP1定位在小麦第5同源群上。对TaABP1-D的克隆和序列比对证实, TaABP1-D与OsABP1同源,
而且不同物种中ABP1基因功能可能非常保守。TaABP1-D在小麦拔节期以叶尖中的表达量最大, 其次是茎上部, 在根和茎基部也可检测到一定水平的表达。在TaABP1-D基因上游开发了一个SSR功能标记, 与株高显著关联。初步推断TaABP1-D基因可能控制株型发育。
References
[1] Vanneste S, Friml J. Auxin: a trigger for change in plant deve-
lopment. Cell, 2009, 136: 1005–1016
[2] Dharmasiri N, Dharmasiri S, Estelle M. The F-box protein TIR1
is an auxin receptor. Nature, 2005, 435: 441–445
[3] Kepinski S, Leyser O. The Arabidopsis F-box protein TIR1 is an
auxin receptor. Nature, 2005, 435: 446-451
[4] Jurado S, Abraham Z, Manzano C, Lopez-Torrejon G, Pacios L F,
Del Pozo J C. The Arabidopsis cell cycle F box protein SKP2A binds to auxin. Plant Cell, 2010, 22: 3891–3904
[5] Renate D, Hartwig L, Bianka S. ABP1, an auxin receptor for fast
responses at the plasma membrane. Plant Signaling & Behavior, 2010, 5: 1–3
[6] Hertel R, Thomson K S, Russo V E A. In-vitro auxin binding to
particulate cell fractions from corn coleoptiles. Planta, 1972, 107: 325–340
[7] Tillmann U, Viola G, Kayser B, Siemeister G, Hesse T, Palme K,
Lobler M, Klambt D. cDNA clones of the auxin-binding protein from corn coleoptiles (Zea mays L.): isolation and characteriza-tion by immunological methods. EMBO J, 1989, 8: 2463–2467 [8] David K M, Couch D, Braun N, Brown S, Grosclaude J, Per-rot-Rechenmann C. The auxin-binding protein 1 is essential for the control of cell cycle. Plant J, 2007, 50: 197–206
[9] Braun N, Wyrzykowska J, Muller P, David K, Couch D, Perrot-Rechenmann C, Fleming A J. Conditional repression of AUXIN BINDING PROTEIN1 reveals that it coordinates cell division and cell expansion during postembryonic shoot deve- lopment in Arabidopsis and tobacco. Plant Cell, 2008, 20: 2746–2762
[10] Perrot-Rechenmann C. Cellular responses to auxin: division ver-sus expansion. Cold Spring Harb Perspect Biol, 2010, 2: a001446
[11] Xu T, Wen M, Nagawa S, Fu Y, Chen J G, Wu M J, Perrot-
Rechenmann C, Friml J, Jones A M, Yang Z. Cell surface- and rho GTPase-based auxin signaling controls cellular interdigita-tion in Arabidopsis. Cell, 2010, 143: 99–110
[12] Robert S, Kleine-Vehn J, Barbez E, Sauer M, Paciorek T, Baster
abp138 小麦生长素结合基因TaABP1-D的克隆、功能标记开发及其与株高的关联
第11期
乔麟轶等: 小麦生长素结合基因TaABP1-D的克隆、功能标记开发及其与株高的关联 2041
P, Vanneste S, Zhang J, Simon S, ?ovanová M, Hayashi K, Dhonukshe P, Yang Z B, Bednarek S Y, Jones A M, Luschnig C, Aniento F, Za?ímalová E, Friml J. ABP1 mediates auxin inhibi-tion of clathrin-dependent endocytosis in Arabidopsis. Cell, 2010, 143: 111–121
[13] Paciorek T, Zaz?malova E, Ruthardt N, Petrasek J, Stierhof Y D,
Kleine-Vehn J, Morris D A, Emans N, Jurgens G, Geldner N, Friml J. Auxin inhibits endocytosis and promotes its own ef?ux from cells. Nature, 2005, 435: 1251–1256
[14] Choi S Y. Molecular cloning and expression of the hot pepper
ERabp1 gene encoding auxin-binding protein. Plant Mol Biol, 1996, 32: 995–997
[15] Huang Y(黄妤), Liu F(刘峰), Guo Q-Q(郭清泉), Zhang X-W(张
学文). Cloning and expression of auxin-binding proteins 1 gene in ramie [Boehmeria nivea (Linn.) Gaud.]. Acta Agron Sin (作物学报), 2008, 34(8): 1358–1365 (in Chinese with English ab-stract)
[16] Devos K M, Gale M D. The use of random amplified polymor-phic DNA marker in wheat. Theor Appl Genet, 1992, 84: 567–572
[17] Gai J-Y(盖钧镒). Method for Experimental Statistics (试验统计
方法). Beijing: China Agriculture Press, 2000. pp 90–91 (in Chinese)
[18] Cechin A L, Sinigaglia M, Lemke N, Echeverrigaray S, Cabrera
O G, Pereira G, Mombach J. Cupin: a candidate molecular struc-ture for the Nep1-like protein family. London: BioMed Central Ltd., 2008 [2012-03-10]. http://www.biomedcentral.com/1471- 2229/8/50
[19] Wang B(王冰), Li J-Y(李家洋), Wang Y-H(王永红). Advances
in understanding the roles of auxin involved in modulating plant architecture. Chin Bull Bot (植物学通报), 2006, 23(5): 443–458 (in Chinese with English abstract)
[20] Friml J, Benkova E, Blilou I, Wisniewska J, Hamann T, Ljung K,
Woody S, Sandberg G, Scheres B, Jurgens G, Palme K. AtPIN4 mediates sink-driven auxin gradients and root patterning in
Arabidopsis. Cell, 2002, 108: 661–673
[21] Benjamins R, Scheres B. Auxin: the looping star in plant devel-opment. Annu Rev Plant Biol, 2008, 59: 443–465
61阅读提醒您本文地址:
[22] Li Y C, Korol A B, Fahim T, Nevo E. Microsatellites within
genes: structure, function, and evolution. Mol Biol Evol, 2004, 21: 991–1007
[23] Chapman M A, Pashley C H, Wenzler J, Hvala J, Tang S X,
Knapp S J, Burkea J M. A genomic scan for selection reveals candidates for genes involved in the evolution of cultivated sun-flower (Helianthus annuus). Plant Cell, 2008, 20: 2931–2945 [24] Hao C-Y(郝晨阳), Dong Y-C(董玉琛), Wang L-F(王兰芬), You
G-X(游光霞), Zhang H-N(张洪娜), Ge H-M(盖红梅), Jia J-Z(贾继增), Zhang X-Y(张学勇). Construction and genetic diversity analysis of common wheat core collection in China. Chin Sci Bull (科学通报), 2008, 53(8): 908–915 (in Chinese without Eng-lish abstract)
[25] Dong Y-C(董玉琛), Cao Y-S(曹永生), Zhang X-Y(张学勇), Liu
S-C(刘三才), Wang L-F(王兰芬), You G-X(游光霞), Pang B-S(庞斌双), Li L-H(李立会), Jia J-Z(贾继增). Development of candidate core collections in Chinese common wheat germplasm. J Plant Gene Resour (植物遗传资源学报), 2003, 4(1): 1–8 (in Chinese with English abstract)
[26] Vigouroux Y, Mitchell S, Matsuoka Y, Hamblin M, Kresovich S,
Smith S, Jaqueth J, Smith O, Doebley J. An analysis of genetic diversity across the maize genome using microsatellites. Genetics, 2005, 169: 1617–1630
[27] Asano K, Yamasaki M, Takuno S, Miura K, Katagiri S, Ito K,
Doi K, Wu J Z, Ebana K, Matsumoto T, Innan H, Kitano H, Ashikari M, Matsuoka M. Artificial selection for a green revolu-tion gene during japonica rice domestication. Proc Natl Acad Sci USA, 2011, 108: 11034–11039
[28] Effendi Y, Rietz S, Fischer U, Scherer G F E. The heterozygous
abp1/ABP1 insertional mutant has defects in functions requiring polar auxin transport and in regulation of early auxin-regulated genes. Plant J, 2011, 65: 282–294
61阅读提醒您本文地址:
五 : 维生素A与肿瘤的关系
对肿瘤的大部分研究是集中在早期发现恶性细胞并毁坏它们,或防止环境中的致癌因素与容易感染的组织接触,但很少有人企图在细胞与致癌因素接触后,以及细胞转变为恶性的阶段如何设法逆转。MaughⅡ报告了美国国立卫生研究院(NIH),以国立癌症研究所(NCI)为主成立了一个研究小组,专门研究维生素A与肿瘤发生的关系,他们认为有效的逆转因子之一是维生素A及其衍生物。
一般肿瘤的发生与成长分三期,即开始,肿瘤前期与形成。肿瘤前期是从开始期向形成期转变,一般在人需要20年,肿瘤前期的细胞如何变化,人们了解不多,但有一点是清楚的,即肿瘤前期的细胞期的细胞会进行修补,并自行恢复正常。维生素A的主要作用是使基因已改变的少数细胞发生逆转。
维生素A的功能之一是使上皮细胞分化成特定的组织。因为肿瘤细胞的发生与上皮细胞分化能力的丧失有关。所以维生素A在防止肿瘤的发生中起了重要作用。上述小组中的很多科学家发现在长期缺乏维生素A的动物中,致癌因子特别能使这些动物发生肿瘤。
NCI的Sporn与Kaufman发现维生素A缺乏的田鼠所分离的气管,在培养液中比正常健康田鼠气管,其DNA与各种致癌因子的结合更紧密。缺乏维生素A动物的肿瘤发生率比不缺乏的发生率要高。麻省理工学院(MIT)的Newberne与Rogers发现缺乏维生素A大鼠,因与致癌因子黄曲霉素B1接触后,大肠癌的发生率比对照显著升高。虽然这些结果还是初步的,但非常重要。
此外,补充维生素A或它的衍生物能预防肿瘤。例如Kaufman发现在田鼠气管的培养液中,加入醋酸视黄酯能抑制致癌因子与DNA的结合,Chopro发现视黄醇及衍生物加入到小鼠前列腺培养液中,能抑制致癌因子的作用,并使致癌作用逆转。
有些研究甚至证明维生素A及它的衍生物对已形成的肿瘤有治疗作用。Swarm发现大鼠移殖软骨肉瘤以后,补充维生素A衍生物(1,3-顺式-视黄醇)能抑制肉瘤生长。Bollag发现视黄醇能使小鼠因致癌因子诱发的乳头瘤的体积缩小。Bollag还发现视黄酸类似物还能使人体的鳞状细胞与基底细胞癌消退。
关于维生素A抑制肿瘤生长的机理了解的很少。有人认为它可能抑制体内活化致癌因子的酶。但Metzler认为维生素A化合物能刺激对抗肿瘤的免疫系统。他发现棕榈酸视黄酯比另一对肿瘤有免疫反应的卡介苗(BGG)能产生强100倍的抗肿瘤作用。因此,有些科学家认为维生素A对肿瘤的防治作用,也许是防治肿瘤问题中最重要的发现之一。但服用大剂量维生素A会产生毒性反应。因此他们建议目前重要的课题是如何合成与筛选没有毒性作用的新维生素A衍生物,来预防与治疗肿瘤。在这一问题未解决之前,不宜过分宣传用维生素A来治疗肿瘤,以免大剂量的服用而引起维生素A过多或中毒。
本文标题:维生素d与钙的关系-如下图,镐京与曲阜的关系是[]A.王都与封国都邑B.均为封国都邑C.王都与郡府D.均为郡府61阅读| 精彩专题| 最新文章| 热门文章| 苏ICP备13036349号-1